Contenidos
Secuencia de shine dalgarno
Secuencia anti shine-dalgarno
Hace más de 30 años, los investigadores descubrieron por primera vez en las bacterias una secuencia de nucleótidos de ARN mensajero (ARNm) que los ribosomas reconocen como señal para saber dónde comenzar la síntesis de proteínas. Hoy en día, los programas informáticos de anotación del genoma aprovechan este hallazgo y lo utilizan para ayudar a identificar la ubicación de los codones de inicio. Dado que estas secuencias, que ahora se denominan secuencias Shine-Dalgarno (SD), siempre se encuentran aguas arriba de los codones de inicio, los programas de anotación las buscan en la región 5′ de estos sitios candidatos. En un análisis exhaustivo de 18 genomas bacterianos, los autores demuestran que cuando se buscan secuencias SD, a veces vale la pena analizar lugares poco probables. Al examinar la región que rodea inmediatamente al codón de inicio de las secuencias SD, los autores identifican muchos genes mal anotados y, al hacerlo, ofrecen un método para ayudar a comprobarlos en futuros proyectos de anotación.
Los métodos basados en la similitud de las secuencias incluyen la búsqueda, aguas arriba de los codones de inicio, de subcadenas de las secuencias SD que tengan al menos tres nucleótidos de longitud [5]. Los errores de identificación pueden surgir de este enfoque por varias razones [6]. No existe un umbral de similitud que pueda delinear claramente las secuencias SD reales de los sitios espurios con un grado significativo, pero bajo, de similitud con la secuencia SD. La falta de certeza ha llevado a una serie de observaciones en las que las secuencias de genes parecen dividirse en dos categorías: las que tienen secuencias SD obvias y las que no. La incapacidad de las técnicas de secuenciación para señalar la ubicación exacta de la secuencia SD plantea un problema porque se cree que su ubicación afecta a la iniciación de la traducción [7-10].
La secuencia shine-dalgarno en eucariotas
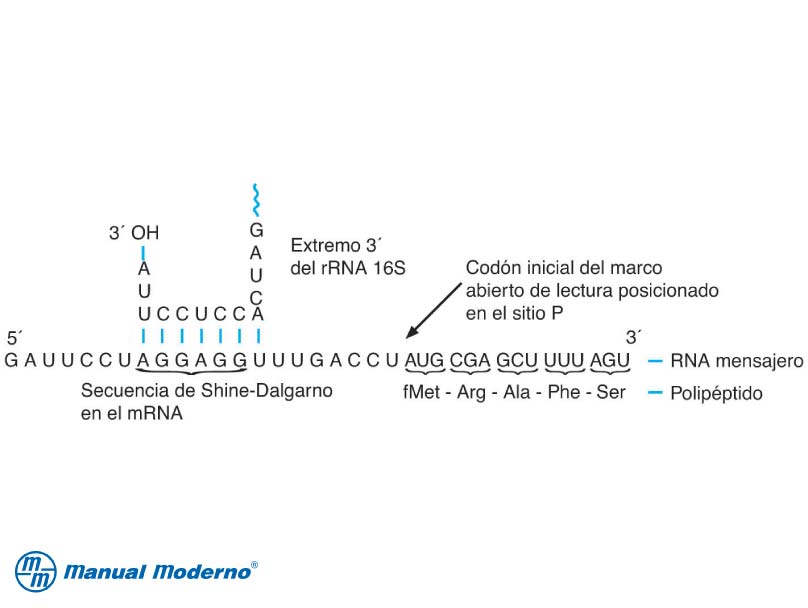
La síntesis de proteínas está regulada por la secuencia y la estructura de la región no traducida (UTR) 5′ del transcrito del ARNm. En procariotas, el sitio de unión al ribosoma (RBS), que promueve la traducción eficiente y precisa del ARNm, se denomina secuencia Shine-Dalgarno en honor a los científicos que la describieron por primera vez. Esta secuencia rica en purinas de la UTR 5′ es complementaria a la secuencia central UCCU del extremo 3′ del ARNr 16S (situado dentro de la subunidad ribosomal pequeña 30S). Se han encontrado varias secuencias Shine-Dalgarno en los ARNm procariotas (véase la figura 1 para la secuencia de consenso). Estas secuencias se encuentran a unos 10 nucleótidos aguas arriba del codón de inicio AUG. La actividad de una RBS puede verse influida por la longitud y la composición nucleotídica del espaciador que separa la RBS y el AUG iniciador.
En los eucariotas, la secuencia Kozak A/GCCACCAUGG, que se encuentra en una corta región no traducida de 5′, dirige la traducción del ARNm. Un ARNm que carezca de la secuencia consenso de Kozak puede traducirse eficazmente en los sistemas in vitro de Ambion si posee una UTR 5′ moderadamente larga que carezca de estructura secundaria estable. Nuestros datos demuestran que, a diferencia del ribosoma de E. coli, que reconoce preferentemente la secuencia Shine-Dalgarno, los ribosomas eucariotas (como los que se encuentran en el lisado reticulado) pueden utilizar de forma eficiente tanto los sitios de unión ribosomal Shine-Dalgarno como Kozak.
La secuencia shine-dalgarno es complementaria a
Se cree que los motivos Shine-Dalgarno (SD) desempeñan un papel importante en la iniciación de la traducción en las bacterias. Paradójicamente, los estudios de perfiles de ribosomas en E. coli no muestran ninguna correlación entre la fuerza del motivo SD de un ARNm y la eficacia de su traducción. Realizando perfiles en ribosomas con secuencias anti-Shine-Dalgarno alteradas, revelamos una correlación en todo el genoma entre la fuerza del SD y la ocupación del ribosoma que anteriormente estaba enmascarada por otros factores contribuyentes. Utilizando el antibiótico retapamulina para atrapar los complejos de iniciación en los codones de inicio, encontramos que los ribosomas mutantes seleccionan correctamente los sitios de inicio, argumentando que los sitios de inicio están preparados para la iniciación a través de la acción de otras características del ARNm. Demostramos que las secuencias ricas en A, aguas arriba de los codones de inicio, promueven la iniciación. En conjunto, nuestro estudio de todo el genoma revela que los motivos SD no son necesarios para que los ribosomas determinen dónde se produce la iniciación, aunque sí afectan a la eficacia de la misma.
Los motivos Shine-Dalgarno tienen la secuencia de consenso GGAGG y pueden emparejarse con hasta nueve nt en la secuencia terminal 3′ del ARNr 16S (ACCUCCUUA en E. coli) denominada anti-Shine Dalgarno o ASD (Shine y Dalgarno, 1974). El emparejamiento de las secuencias SD-ASD puede reclutar subunidades 30S al codón de inicio 5-10 nt aguas abajo (Steitz y Jakes, 1975). Los motivos SD que difieren significativamente del consenso o que están posicionados demasiado cerca o demasiado lejos del codón de inicio producen niveles más bajos de iniciación. De hecho, muchos experimentos utilizando genes reporteros mostraron que el aumento de la afinidad SD-ASD aumenta la producción de proteínas, demostrando su importancia para determinar los niveles de traducción (Hui y de Boer, 1987; Jacob et al., 1987; de Smit y van Duin, 1990; Salis et al., 2009). Además, el modelo de DS sirve de base para los esfuerzos prácticos de bioingeniería que van desde la optimización de la expre
Función de la secuencia shine-dalgarno
La secuencia Shine-Dalgarno (o caja Shine-Dalgarno), propuesta por los científicos australianos John Shine y Lynn Dalgarno,[1] es un sitio de unión ribosómica situado generalmente 6-7 nucleótidos antes del codón de inicio AUG. La secuencia Shine-Dalgarno sólo existe en procariotas; la secuencia consenso de seis bases es AGGAGG. Esta secuencia ayuda a reclutar el ribosoma hacia el ARNm para iniciar la síntesis de proteínas, alineándolo con el codón de inicio. La secuencia complementaria (CCUCCU), se denomina secuencia anti-Shine-Dalgarno y se encuentra en el extremo 3′ del ARNr 16S en el ribosoma. El equivalente eucariótico de la secuencia Shine-Dalgarno se denomina secuencia Kozak. La secuencia Shine-Dalgarno en E. coli, por ejemplo, es AGGAGGA.
Las mutaciones en la secuencia Shine-Dalgarno pueden reducir la traducción. Esta reducción se debe a una menor eficiencia de emparejamiento ARNm-ribosoma, como demuestra el hecho de que las mutaciones complementarias en la secuencia anti-Shine-Dalgarno pueden restaurar la traducción.
Cuando la secuencia Shine-Dalgarno y la secuencia anti-Shine-Dalgarno se emparejan, los factores de iniciación de la traducción IF2-GTP, IF1, IF3, así como el ARNt iniciador fMet-tRNA(fMET) son reclutados al ribosoma.